Bestimmung der thermischen Stabilität (Tm) von Proteinen mittels nanoDSF

BioDevCenter
Teamleiterin F&E
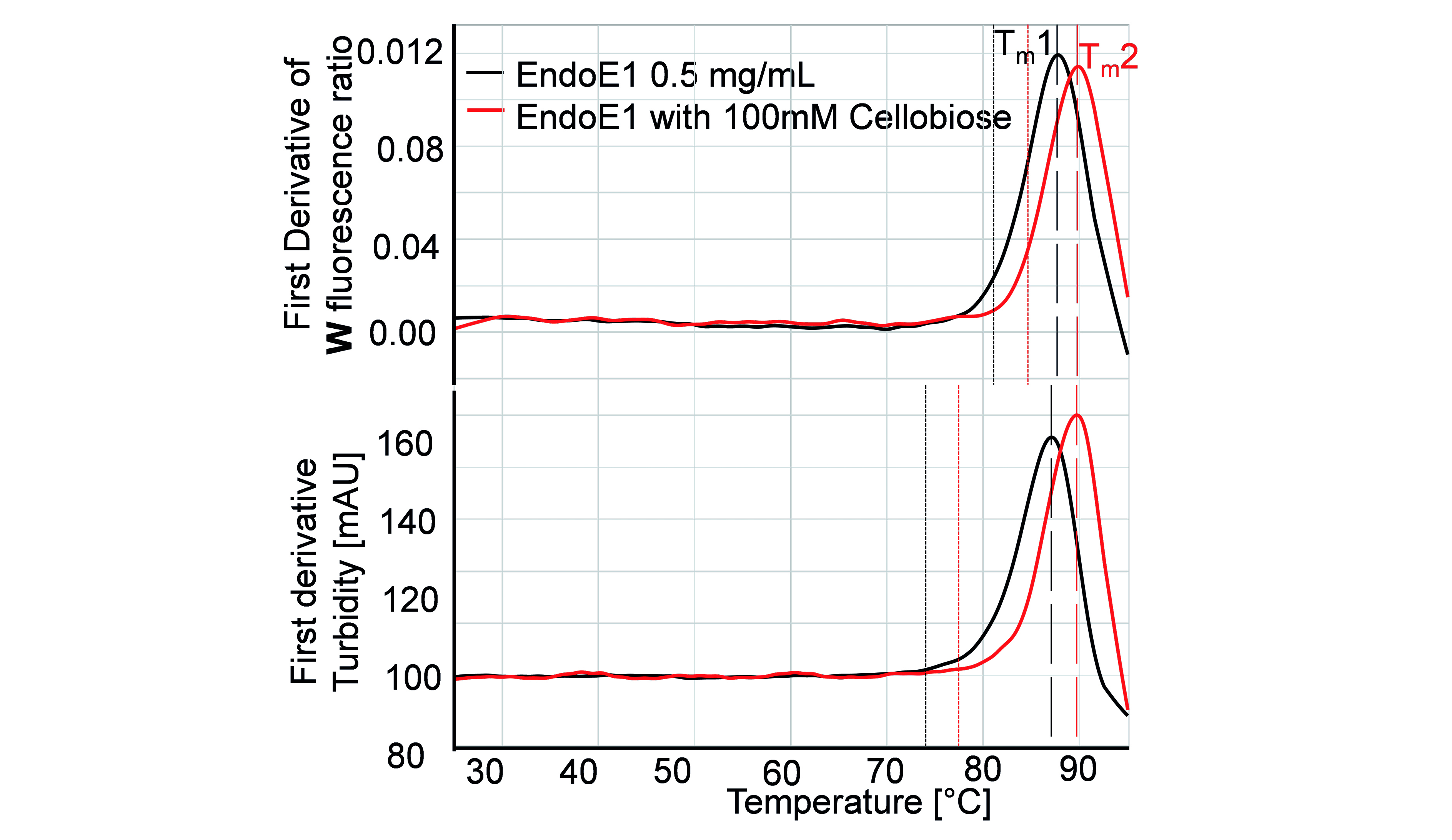
Die thermische Stabilität ist ein wichtiger Parameter bei der Auswahl von geeigneten Enzymen, Antikörpern oder anderen biotechnologisch hergestellten Proteinen für industrielle oder pharmazeutische Anwendungen. Das NMI bietet die Bestimmung des Protein-„Schmelzpunktes“ Tm im Screeningverfahren an. Hierfür wird die nanoDSF-Technologie genutzt, welche auf Änderungen der Tryptophanfluoreszenz in Abhängigkeit der molekularen Umgebung beruht.
Eine korrekte dreidimensionale Faltung ist unerlässlich für die biologische Funktion von Proteinen als Katalysatoren oder als Bindungspartner für andere Moleküle.
Die Stabilität der dreidimensionalen Anordnung ist eine intrinsische Moleküleigenschaft. Sie ist abhängig von intramolekularen Bindungen und Wechselwirkungen, wie z.B. Disulfidbrücken, ionischen oder Wasserstoffbrücken-Bindungen, von der primären Abfolge der Aminosäuren und von der Art der posttranslationalen Modifikationen, wie z.B. Glykosylierungen. Gibt es mehrere Kandidaten für ein Zielproduktprofil (target product profile) oder erhöhte Anforderungen an die thermische Stabilität, so kann mittels nanoDSF im Screeningverfahren der geeignetste Kandidat schnell und mit wenig Materialbedarf ermittelt werden.
Die dreidimensionale Stabilität ist jedoch zusätzlich abhängig von Umgebungsbedingungen, wie z.B. pH-Wert und Pufferzusammensetzung oder Anwesenheit von anderen Molekülen wie z.B. Bindungspartnern, Lösungsmitteln oder chaotropischen Reagenzien. Mittels nanoDSF können am NMI für ein bestimmtes Zielmolekül verschiedene Formulierungen getestet werden und so z.B. optimale Lagerpuffer, Reaktionsbedingungen oder Resistenzen gegenüber destabilisierenden Bedingungen gefunden werden.
Gerne erstellen wir ein maßgeschneidertes Angebot für Sie!
Hier finden Sie interessante Poster zu dieser Dienstleistung mit weiteren Informationen: Analytical Characterization of Commercial Recombinant Endoglucanase E1, Optimizing reaction conditions for deglycosylation of intact proteins using a novel enzyme PNGase Rc